목차
선형회귀모형에서 제일 많이 사용되는 bootstrap은 paired bootsrap이다. 이는 data table이 있다면 row를 resampling하는 방식이다. 즉, $(X_i, Y_i)$를 pair로 resampling하는 것이다.
이 방식은 단순하기 때문에 실행하기 쉽다는 장점이 있다. 하지만 influential points가 있는 경우, 크게 영향을 받는다는 단점이 있다. 이는 resampling 과정에서 influential points가 샘플링 되는지에 따라 추정치가 크게 변하기 때문이다.
이 포스트에서는 influential points에 영향을 덜 받는 residual bootstrap을 알아보자.
1. Algorithm
먼저, 가장 단순한 선형회귀 분석의 경우를 보자. 데이터 $\{(X_i, Y_i)\}_{i=1}^{n}$가 주어졌다고 가장하자. 그리고 다음과 같은 선형회귀식을 가정하자.
$$Y_i = \alpha + \beta X_i + \epsilon_i$$ where $\epsilon_i \sim iid(0,\sigma)$
잔차(residual)를 활용한 bootstap의 알고리즘은 다음과 같다.
1. $\alpha$와 $\beta$를 추정한다. 이 추정치를 $\hat{\alpha}$와 $\hat{\beta}$라고 하자.
2. $\hat{\alpha}$와 $\hat{\beta}$를 이용하여 잔차를 구한다. $\hat{\epsilon}_i = Y_i - \hat{\alpha} - \hat{\beta}X_i$
3. 잔차를 centering 해준다. $e_i = \hat{\epsilon}_i - \bar{\hat{\epsilon}}$. 이는 bias를 줄이기 위함이다.
4. $e_i$를 resampling해서 bootstrap residual $e^*_1,...,e^*_n$을 얻는다.
5. 다음과 같이 bootstrap sample $\{(X^*_i, Y^*_i)\}_{i=1}^{n}$을 얻는다.
$$X^*_i = X_i, Y^*_i = \hat{\alpha} + \hat{\beta}X_i + e^*_i$$
1~5의 과정을 수행하면 1개의 bootstrap sample을 얻을 수 있다.
2. Simulation
이제 R로 시뮬레이션을 해보자. 다음과 같은 모델을 가정한다.
$$Y_i = 3 + 2X_i + \epsilon_i$$
여기서 오차항 $\epsilon_i $ 는 95% 확률로 표준정규분포를 따르고, 5%의 확률로 표준편차가 8인 정규분포를 따른도록 한다.
표본의 크기 $n$은 31로 설정하자.
2-1. Data generating process
Data generating process는 다음과 같이 하자. x는 0~15 사이의 숫자를 sampling 하고, y는 가정한 모델을 따라 만든다.
## Data generating process
dgp_x <- function(n=31){
set.seed(112815)
x = sample(0:15, size = n, replace = TRUE)
return(x)
}
dgp_yx <- function(x, k=8, a=3, b=2){
n <- length(x)
e <- rnorm(n, 0, (1 + (k - 1)*rbinom(n, 1, 0.05))) ## error
y <- a+ b*x + e
return(cbind(y,x))
}
추정량으로는 Least Absolute Deviation (LAD) estimator와 최소제곱 (LS)을 사용해보자.
추정에 앞서 결과를 예상해보자:
1. 오차항이 정규분포를 따르지 않기 때문에 LS의 standard error 공식은 틀리게 된다.
2. LS는 outlier에 민감하지만, LAD는 그렇지 않다.
따라서, 전반적인 performance는 LAD가 좋을 것이라고 예상할 수 있다.
2-2. LAD estimator
그럼 이제 LAD 추정치를 return하는 함수를 정의하자.
#LAD estimate
lad <- function(data){
est.lad <- nlminb(coef(lm(data[,1]~data[,2])),
function(x){sum(abs(data[,1] - (x[1] + x[2]*data[,2])))})
return(est.lad$par)
}
2-3. Standard error of LAD
LAD는 closed-form이 존재하지 않기 때문에 공식을 통해서 standard error를 구할 수 없다. 여기서 bootstrap이 등장할 차례이다.
Bootstrap sample을 얻고, bootstrap LAD 추정치를 얻는 과정을 1000번 반복한다. 1000개의 bootstrap LAD 추정치는 주어진 데이터의 LAD 추정량의 대략적인 분포를 나타낸다. LAD의 standard error는 1000개의 bootstrap LAD 추정치의 sample standard deviation이 된다.
lad.est <- function(data, R=1000){
est = lad(data) # get estimation of a and b
fit.y = est[1] + est[2]*data[,2]
e = data[,1] - fit.y
e = e - mean(e)
bt.star <- function(data, e, est){
e = sample(e, replace = TRUE)
y.star = est[1]+ est[2]*data[,2] + e
return(lad(cbind(y.star, data[,2])))
}
output = replicate(R, bt.star(data, e, est))
se.a = sd(output[1,])
se.b = sd(output[2,])
return (c(est, se.a, se.b))
}
아래의 함수는 LAD와 LS의 결과를 종합하여 return하는 함수이다.
one_result <- function(data, R=1000){
lm.est <- lm(data[,1]~data[,2])
return(c(lad.est(data, R),
coef(lm.est),
sqrt(diag(vcov(lm.est)))))
}
2-4. Result
이제 parallel computing을 통해 Monte Carlo simulation을 해보자. 시뮬레이션 횟수는 1000번이다.
library(parallel)
x <- dgp_x()
cl <- makeCluster(detectCores())
clusterExport(cl, "x")
clusterExport(cl, "dgp_yx")
clusterExport(cl, "lad")
clusterExport(cl, "lad.est")
clusterExport(cl, "one_result")
clusterSetRNGStream(cl, iseed=215333)
para <- parApply(cl, replicate(1000,dgp_yx(x)), 3, one_result, R=1000)
stopCluster(cl)결과를 예쁘게 plot으로 정리하면 다음과 같다.
result <- as.data.frame(t(para))
colnames(result) <- c("LAD.a", "LAD.b", "LAD.se.a", "LAD.se.b",
"LS.a", "LS.b", "LS.se.a", "LS.se.b")
library(ggplot2)
library(grid)
library(gridExtra)
p1 <- ggplot() + geom_density(aes(x=result$LAD.a, colour="LAD")) +
geom_density(aes(x=result$LS.a, colour="LS")) +
xlab("est.a") + ylab("Density") + ggtitle('est.a density') +
theme(legend.position = 'right') +
scale_color_manual(values = c('LAD' = 'red', 'LS' = 'blue'))
p2 <- ggplot() + geom_density(aes(x=result$LAD.b, colour="LAD")) +
geom_density(aes(x=result$LS.b, colour="LS")) +
xlab("est.b") + ylab("Density") + ggtitle('est.b density') +
theme(legend.position = 'right') +
scale_color_manual(values = c('LAD' = 'red', 'LS' = 'blue'))
p3 <- ggplot() + geom_density(aes(x=result$LAD.se.a, colour="LAD")) +
geom_density(aes(x=result$LS.se.a, colour="LS")) +
xlab("se.a") + ylab("Density") + ggtitle('se.a density') +
theme(legend.position = 'right') +
scale_color_manual(values = c('LAD' = 'red', 'LS' = 'blue'))
p4 <- ggplot() + geom_density(aes(x=result$LAD.se.b, colour="LAD")) +
geom_density(aes(x=result$LS.se.b, colour="LS")) +
xlab("se.b") + ylab("Density") + ggtitle('se.b density') +
theme(legend.position = 'right') +
scale_color_manual(values = c('LAD' = 'red', 'LS' = 'blue'))
grid.arrange(p1, p2, p3, p4, ncol=2,
top = textGrob("k= 8", gp=gpar(fontsize=20,font=3)))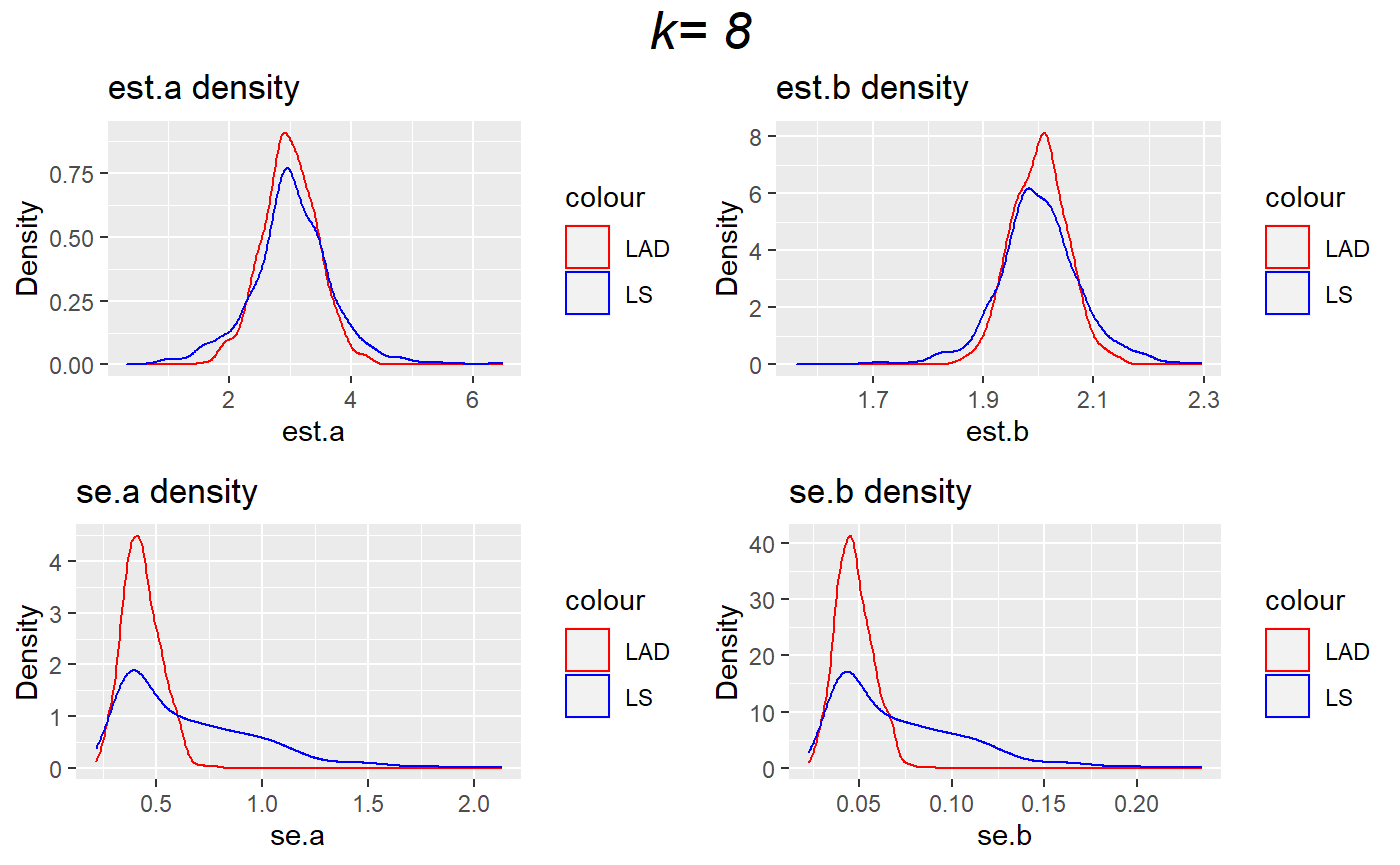
결과는 우리가 예상했대로이다.
1. 위의 plot들은 시뮬레이션한 각 추정량의 분포를 나타낸다. LAD와 LS 모두 불편성을 만족하지만, LAD 추정량의 분산이 LS보다 작다. 즉, 우리의 모델에서는 LAD가 더 효율적인 추정량이다.
2. 아래의 plot들은 시뮬레이션한 각 추정량의 standard error의 분포를 나타낸다. LAD의 standard error의 분산이 LS보다 작음을 확인할 수 있다.
이번엔 standard error에 더 집중해보자.
simulation을 통해 얻은 1000개의 추정량의 분포는 실제 추정량의 분포에 가까울 것이다. 따라서, 우리가 얻은 분포의 표준편차는 실제 분포의 표준편차 값에 가까울 것이다. 또한 추정량이 nicely perform한다면, 표준편차 값은 standard error의 평균에 가까울 것을 예상할 수 있다.
이제 plot을 그려서 확인해보자.
p1 <- ggplot() + geom_density(aes(x=result$LAD.se.b, colour="se")) +
xlab("LAD.se.b") + ylab("Density") + ggtitle('Density of LAD.se.b') +
geom_vline(aes(xintercept=sd(result$LAD.b), colour='true')) +
theme(legend.position = 'right') +
scale_color_manual(values = c('LAD.se.b' = 'black', 'true' = 'blue'))
p2 <- ggplot() + geom_density(aes(x=result$LS.se.b, colour="se")) +
xlab("LS.se.b") + ylab("Density") + ggtitle('Density of LS.se.b') +
geom_vline(aes(xintercept=sd(result$LS.b), colour='true')) +
theme(legend.position = 'right') +
scale_color_manual(values = c('LS.se.b' = 'black', 'true' = 'blue'))
grid.arrange(p1, p2, ncol=1,
top = textGrob("k= 8", gp=gpar(fontsize=20,font=3)))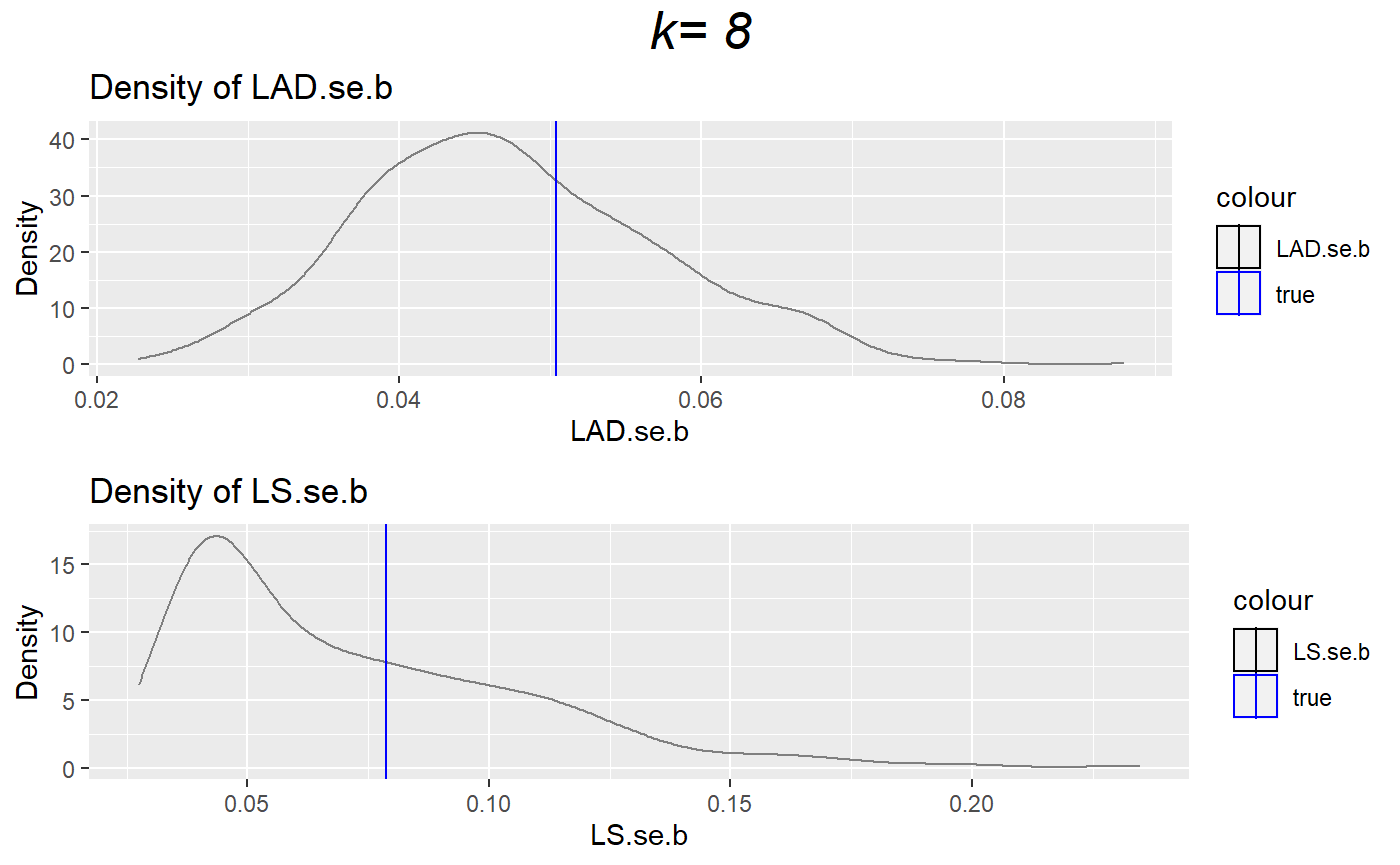
각 plot의 파란 선은 시뮬레이션을 통해 얻은 1000개의 추정치의 표준편차 값이다. LAD의 경우, 파란 선이 분포의 중심에 가까운 것을 확인할 수 있다. 하지만 LS의 경우, 파란 선이 분포의 중심에서 멀리 떨어져있다. 이는 LS의 standard error가 좋지 않음을 나타낸다. (오차항이 정규분포를 따르지 않기 때문이다.)
지금까지 residual bootstrap의 절차와 시뮬레이션을 통한 예시를 봤다. 다음 포스트에서는 bootstrap으로 신뢰구간을 구하는 것을 다뤄보고자 한다.
 Skypainter
Skypainter